学术资源
X-CNV:全基因组预测拷贝数变异的致病性

作者开发了一款新的计算框架(X-CNV)用于CNVs致病性的预测。该方法使用XGBoost算法,整合了超过30个信息特征,在训练集和验证集中的AUC值分别能达到0.96和0.94,优于现有的其它致病性评估工具。作者还基于XGBoost算法生成的概率值建立了meta-voting prediction (MVP)评分来定量衡量致病效应。该评分在不同种族人群中对遗传性状/疾病的致病CNVs有较高的鉴别力。
背景介绍
现有预测CNV致病性的方法可以分为3类:
整合CNV区间的每个单核苷酸致病性评分,来确定CNV的致病性。比如SVscore。
基于规则策略,对CNV的致病性进行优先级排序。如ACMG,ClinGen指南。
基于的单倍剂量不足预测来评估CNV的影响。
X-CNV通过集成各种公开数据库,整合了最全面的CNV数据和注释。使用的是机器学习的方法( XGBoost algorithm),为了提高预测能力,将基因组学、基因组区域、变异类型和群体遗传学等信息特征纳入其中。基于XGBoost算法生成的概率值,建立了meta-voting prediction (MVP)评分来定量衡量致病效应。

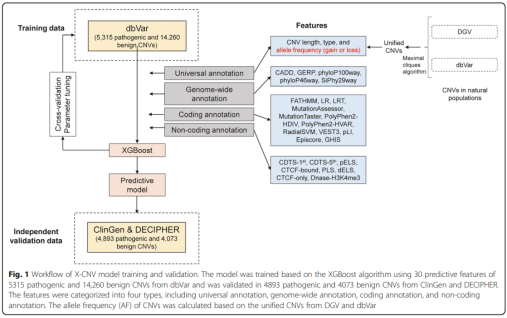
研究内容
数据整理
作者从DGV和dbVar收集了14076147个CNVs,去除相同的坐标位置,得到来自87935个样本的11788451个CNVs。这些样本被分成9个种群。所有的CNV进行合并,去除10MB以上的CNVs,最终得到5315个致病和14260个良性CNVs用于模型开发,4893个致病和4073个良性CNVs用于模型验证。
CNV合并
为了合并来自不同平台不同生信流程的潜在一致的CNV,作者开发了一种基于极大团算法的新型策略来识别具有相近坐标的CNVs。
先将CNV根据染色体进行分组,然后对于每条染色体上的任意两个CNVs之间的距离进行计算:![]()
式中Si、Ei为第i个CNV的起始位置和结束位置,Di,j为CNVi与CNV j之间的距离。
将距离的阈值设为100bp,然后将Di,j转化为二进制标签: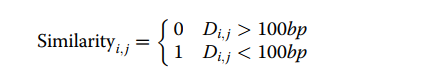
将二进制相似度标签等于1的CNV连接起来,构造一个无向CNV网络。CNV网络由一个或多个子网组成。如果在一个子网内的CNVs是完全连接的,则表示它们是相同的。
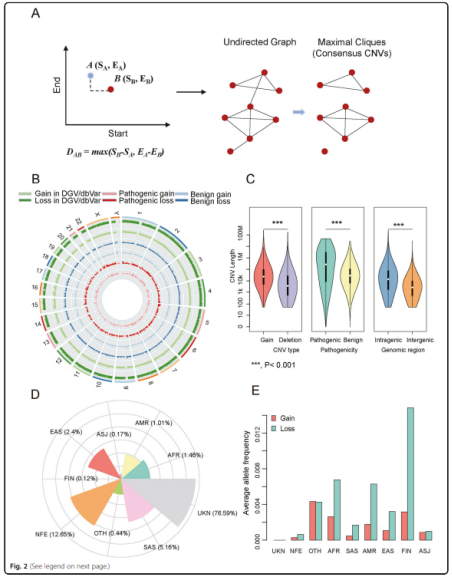
作者对合并后的CNV在自然人群中的通用特征进行了统计。CNVs覆盖了93.7%以上的常染色体(Y染色体除外),这表明CNVs是人类参考基因组中普遍存在的遗传变异。此外,在CNV长度方面,gain 比 deletion长,pathogenic 比 benign长,intragenic 比 intergenic长。
这些CNVs中,种群NFE,SAS,EAS,AFR,AMR占了1%以上。在9个种群中,基因缺失的平均等位基因频率始终高于基因增加的平均等位基因频率。
模型构建
作者从DGV和dbVar收集了14076147个CNVs,去除相同的坐标位置,得到来自87935个样本的11788451个CNVs。这些样本被分成9个种群。所有的CNV进行合并,去除10MB以上的CNVs,最终得到5315个致病和14260个良性CNVs用于模型开发,4893个致病和4073个良性CNVs用于模型验证。
由于LP、VOUS、LB的类别通常是不明确的,作者只使用了被注释为致病和良性的CNVs来建立模型。X-CNV总共使用了30个预测特征,分为四种类型:通用特征(4个)、编码区特征(13个)、非编码区特征(8个)和全基因组特征(5个)。包括突变水平和基因水平。
X-CNV使用XGBoost分类器来区分致病性和良性CNV。使用100次10倍交叉验证优化XGBoost的超级参数。采用gbtree booster和logistic回归的XGBoost模型获得了最高的AUC(0.9740±0.0058),性能最佳。使用优化后的超级参数对整个训练集进行了XGBoost模型的开发,得到的AUC值为0.96。然后使用独立的验证集对开发好的模型进行验证。

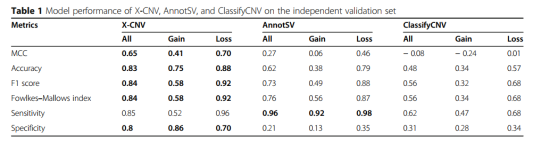
作者将X-CNV与其他结构变异致病性评估软件进行比较,X-CNV获得了最高的AUC值(0.94),与SVScores相比提高了3.5% ~ 33.7%。

作者使用6个性能指标对几款软件的效果进行比较。除了灵敏度以外,X-CNV在其他5个指标中的表现都优于其他两款软件(AnnotSV ,ClassifyCNV)。虽然AnnotSV获得了最高的灵敏度,但是同时特异性是最低的,说明AnnotSV更趋于把CNV预测为P或者LP。X-CNV提供了最平衡的敏感性和特异性,显示其在区分假阳性和阴性时的优越能力。
X-CNV的重要特征
作者从优化后的XGBoost模型中提取了前10个预测特征。
前10名最具预测价值的特征包括4个编码区特征(LR score, VEST3 score, FATHMM score , pLI)、3个通用特征(CNV type, length, loss-PAF)、2个非编码区特征(PLS和CTCF−bound)和1个全基因组特征(CADD)。LR评分对XGBoost模型的性能贡献在50%以上。
将验证集的CNVs分为0~10 kb、10 kb ~ 100 kb、100 kb ~1 Mb和1 Mb ~10 Mb 4组。4组的AUC均在0.85以上,说明X-CNV模型在不同长度的CNV中都能达到较高的性能,但CNV长度越长预测的精度越高。X-CNV模型对CNV缺失的AUC(0.92)高于重复的AUC(0.89)。

MVP评分定量测定CNV致病性
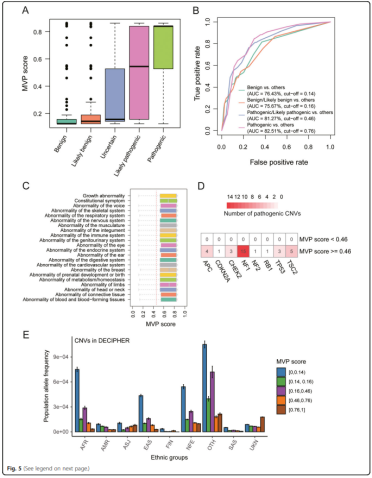
为了定量测定CNV与致病性之间的关系,以支持潜在的临床应用,作者基于XGBoost算法生成的概率值,开发了MVP评分。X-CNV的概率值能够区分不同的致病效应,具有统计学意义。使用ROC-AUC分析确定每个致病性分类的最佳概率截断值。
X-CNV的应用
作者基于HPO将DECIPHER中的1666 CNVs分类到22种罕见疾病类型,使用X-CNV进行致病性预测,其中85%都被预测为LP/P。所有罕见病类型的MVP得分的25%分位数均大于0.46,提示CNVs可能被X-CNV预测为致病或可能致病的。
为了验证X-CNV在癌症易感基因(CPGs)中的应用,作者使用了DECIPHER中具有明确表型并且位于抑癌基因区的32个CNV缺失,所有的CNVs都被预测为LP/P。
作者为了验证X-CNV在群体遗传中的应用,进一步研究了X-CNV在验证集的9个民族中区分CNV致病性的能力。验证集中致病性CNVs 的人群频率比良性CNVs的人群频率低得多。这与表观遗传学的研究结果一致,即由于净化选择,致病性变异在健康人群中极为罕见。
研究结论
综上所述,X-CNV可以在全基因组范围内定量地区分功能性、有害和致病的CNV,在群体遗传学、疾病相关性研究和诊断筛选方面具有广泛的应用价值。
X-CNV下载地址:
Web server:www.unimd.org/XCNV
Github地址:https://github.com/kbvstmd/XCNV
参考文献:
Zhang L., Shi J., Ouyang J., Zhang R., Tao Y., Yuan D., Lv C., Wang R., Ning B., Roberts R., et al. X-CNV: Genome-Wide Prediction of the Pathogenicity of Copy Number Variations. Genome Med. 2021;13:132. doi: 10.1186/s13073-021-00945-4.
