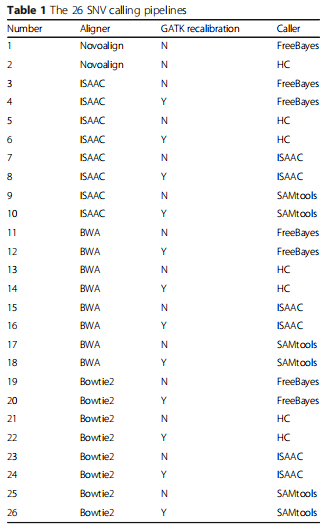
学术资源
参考基因组hg19和hg38间的变异检测结果异同
参考基因组的选择对于NGS数据的分析至关重要,目前最常用的参考基因组版本为hg19和hg38。该篇文献详细评估了参考基因组hg19和hg38版本对SNV检出的影响。
方法
文献利用GIAB项目中NA12878的全基因组测序(WGS)数据,分别基于参考基因组hg19和hg38通过26个pipepline进行SNV calling。之后分别使用转换工具Picard和CrossMap,对hg19和hg38的SNVs进行相互转换,并统计了hg19和hg38的相互转换率,以及成功转换但不一致的SNV比例,最后对不一致的原因进行了分析。
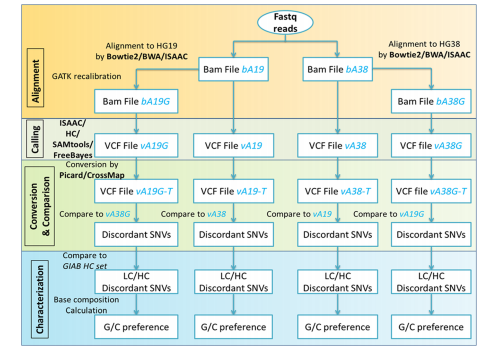
文献使用了三款比对软件Bowtie2、BWA和ISAAC,并研究了GATK重比对是否对结果产生影响(图1)。结果表明使用不同版本的基因组对整体比对率影响不大,但是从覆盖度来讲,hg38版本比hg19效果要好且使用hg38比hg19检出更多的SNV(图2)。Bowtie2和BWA比对效果相当,ISAAC在低覆盖区域较其他两款软件有差别。是否进行GATK重比对对结果影响不大。
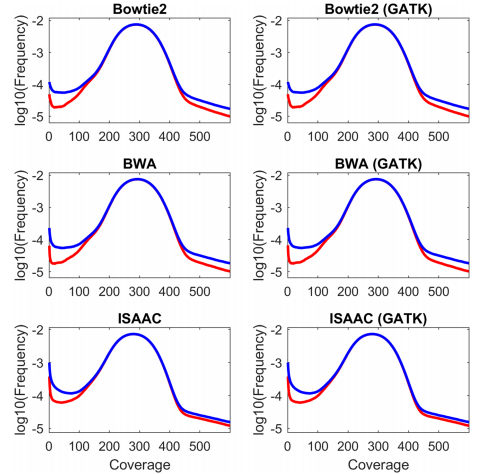
图1
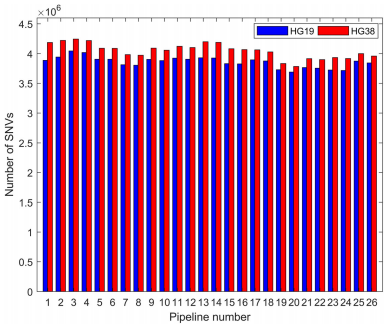
图2
结果
hg19 to hg38的转换率为99%,hg38 to hg19的转换率为95%。说明低版本的基因组坐标很容易转换为高版本,而高版本的坐标则不推荐转到低版本。
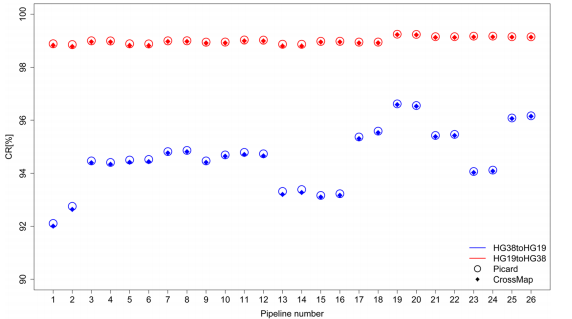
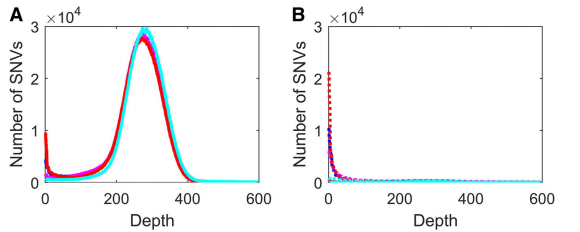
Picard和CrossMap这两款转换软件的表现相当,文献对转换失败的SNV分析发现大多数无法转换的SNV的测序深度非常低。因为基因组中的某些重复序列区域,比对质量值低,从而导致测序深度低。
不同基因组版本互相转换中约有1.5%成功转换的SNV和目标版本检出的SNV结果不一致。
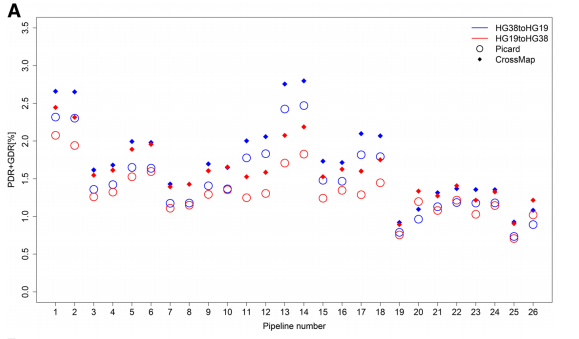
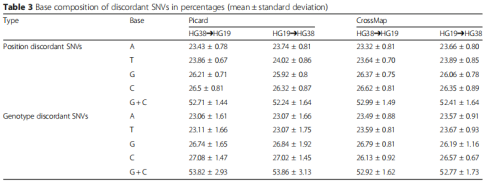
转换不一致的SNV包括位置不一致(PD)和基因型不一致(GD),其中位置不一致的数量远远大于基因型不一致的。
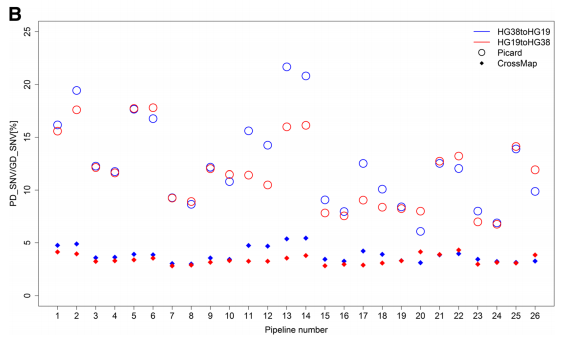
比对软件的选择,也会对基因组版本转换产生影响。三款比对软件中,使用Bowtie2的pipeline有最低的不一致率,而使用BWA的pipeline则产生最高的不一致率。两款转换软件CrossMap 和Picard 相比,CrossMap产生更多的基因型不一致,因而在进行不同基因组版本转换时,建议使用Picard。
文献分析了不同版本基因组相互转换时产生PD(图3) 和GD(图4)的原因。根据GIAB的金标准将PD和GD 的SNV划为高置信(HC)和低置信(LC)SNV, hg38 to hg19产生位置不一致的SNV主要来源于低置信的SNV。SNV置信高低对基因型不一致的产生影响不大。

图3
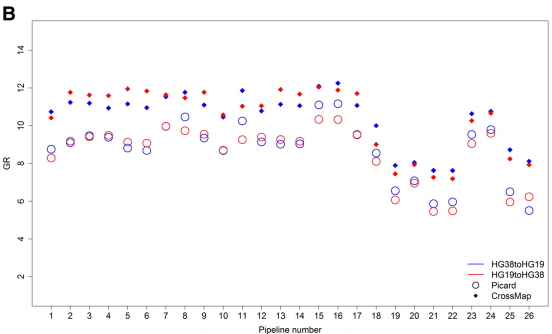
图4
众所周知,GC含量会影响SNV的检出,文献研究发现转换不一致的SNV的GC比例在52.24% -53.86%,明显高于42%的正常GC比例。
讨论
转换软件推荐使用Picard。
参考基因组推荐使用高版本的hg38
不同版本基因组相互转换时,会出现转换失败或不一致,高版本转到低版本失败率和不一致率更高,所以不建议高版本转到低版本。
低置信度或高GC区域的SNV更容易转换后不一致。
