学术资源
ClinGen SVI剪接小组的建议:基于 ACMG/AMP框架,剪接相关的预测性或观察性的证据的使用

简介
ClinGen SVI剪接小组在本文中针对六个与剪接相关的证据类别:PVS1、PS3、PP3、BS3、BP4和BP7证据(以及PS1)在剪接变异分类中的使用提供了统一的建议。
PART 1
根据基序(motif)位置将剪接变异分为3类(图1):
剪接供体/受体±1,2二核苷酸变异
位于内含子5‘或3’端2个碱基区域内。
剪接区变异
发生在供体基序或受体基序内。
最小剪接区变异
发生在上述供体基序,或最小高度保守的受体基序内。
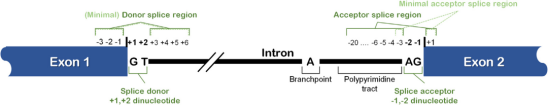
图1
Pre-mRNA的剪接主要依赖于以下顺式作用的调控元件:1.剪接供体;2.剪接受体;3.剪接受体上游的多聚嘧啶序列和分支位点(通常位于-18 ~ -44位置)。
绝大多数内含子(>98%)5'端是GT,3'端是AG,是高度保守的二核苷酸,由U2剪接体(spliceosome)识别。
Burge等(1999)定义了供体/受体基序的边界——供体剪接位点基序:外显子最后面的3个碱基~内含子前8个碱基;受体剪接位点基序:内含子最后面的12碱基~外显子的前2个碱基。
剪接小组对U2型内含子的位置权重矩阵图进行分析,发现这些基序中某些位置有简并性——位于供体基序和受体基序的变异最有可能对剪接产生影响。二次分析后,剪接小组定义了一个最小剪接区域,和上述供体基序一致,但受体剪接区域则缩小为外显子的第1个碱基和外显子边界上游的3个碱基。
PART 2
针对剪接供体/受体±1,2二核苷酸变异的基因特异性PVS1决策树(例举说明)(图 2):
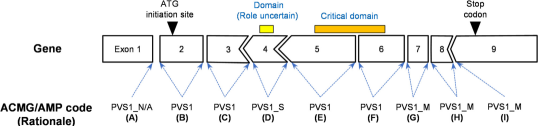
图2
A.PVS1_N/A
5'UTR区域:没有剪接改变预测或使用隐匿剪接基序不影响编码序列。
B.PVS1
外显子跳跃或使用隐匿剪接基序破坏了起始密码子,并且没有可替代的起始密码子。
C.PVS1
外显子跳跃或使用隐匿剪接基序破坏阅读框,并预计会发生NMD。
D.PVS1_S
外显子跳跃或使用隐匿剪接基序保留了阅读框,但丢失了一段蛋白质序列(>10%),该序列未被证实为蛋白关键功能域。
E.PVS1
外显子跳跃或使用隐匿剪接基序会破坏阅读框,并预计会发生NMD。
F.PVS1
外显子跳跃或使用隐匿剪接基序保留了阅读框,并丢失了一个已确定的蛋白关键功能域。
G.PVS1_M
外显子跳跃或使用隐匿剪接基序保留阅读框,但丢失了一段蛋白质序列(<10%),该序列未被证实为蛋白关键功能域。
H.IPVS1_M
外显子跳跃或使用隐匿剪接基序会破坏阅读框,预计不会发生NMD,丢失了一段蛋白质序列(<10%),该序列未被证实为蛋白关键功能域。
注意:如果一个基因既可通过LoF(Loss of Function)又可通过GoF(Gain of Function)机制致病,而该基因符合PVS1条件的变异预计导致GoF,剪接小组建议使用PM4。
PART 3
PVS1/BP7与体外剪接检测数据
对于剪接供体/受体±1,2 二核苷酸变异,当预测结果与体外剪接检测结果不同时,应考虑根据剪接检测结果适当调整PVS1强度;
对于剪接供体/受体 ±1,2 二核苷酸之外的变异。如果体外剪接检测结果提示变异导致异常剪接,也可根据PVS1决策树使用PVS1。
如果PVS1的应用不仅是由于预测结果,还使用了剪接检测数据,建议添加额外注释,如PVS1_Strength(RNA),或注明PVS1的应用是由于剪接检测数据的存在。
建议PS3/BS3仅用于评估功能影响实验。体外剪接检测证实同义变异和内含子变异对剪接没有影响的情况,建议使用BP7_Strong(RNA),或说明BP7_Strong的应用是由于剪接检测数据的支持。
PART 4
根据样本类型、检测技术和转录本复杂性调整证据强度
保守建议PVS1和BP7证据仅适用于非肿瘤组织样本进行的剪接检测结果;
在没有临床数据对实验系统进行校准的情况下,对基于质粒构建的检测技术,如minigene、 MPRA等,建议仅使用较低强度;
对于不完全剪接变异(''leaky''):减少但不完全破坏参考转录本正常剪接的变异,需要确定异常转录本在特定基因-疾病关系中的致病比例。
PART 5
PP3/BP4与剪接预测结果:
对比11种剪接预测工具,剪接小组最终选取SpliceAI展示了测定剪接预测工具阈值的模型(图3)。

图3
最终确定了供体/受体±1,2二核苷酸之外的变异使用PP3的最佳阈值为≥0.2,这一阈值灵敏度约78%。使用BP4的最佳阈值为≤0.1,特异性为87%(图4)。
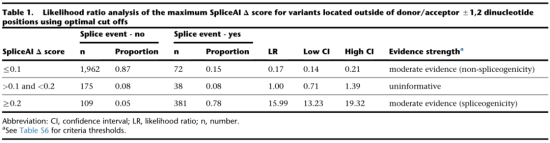
图4
对位于供体/受体±1,2二核苷酸位置以外的变异(图5),
▲SpliceAI评分≤0.1,可使用BP4证据;如果是同义变异或内含子变异(位于供体/受体外);可进一步使用BP7证据。
▲ SpliceAI评分为≥0.2,预测会发生剪接,使用PP3证据;
▲ SpliceAI评分在0.1~0.2之间,考虑为错义变异或Indel。
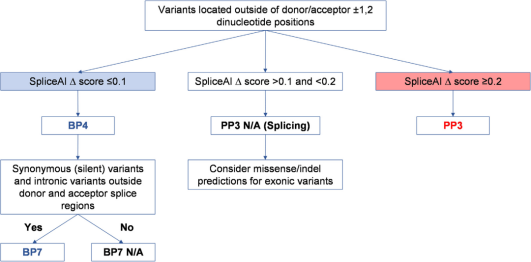
图5
PART 6
BP7的应用:
同义变异,且剪接预测既不会影响剪接,也不会产生新的剪接位点,同时该核苷酸的保守性不高。
1.建议BP7不应用于位于外显子第1个碱基或最后3个碱基的同义替换。
2.建议不要将进化保守性纳入考虑。
3.剪接小组认为可将BP7的应用范围扩大到位于最小剪接区之外的内含子变异。
PART 7
PS1/PM5与剪接预测结果
PM5不适合直接用于剪接变异。
PS1的适用情况需考虑:待分类变异和已知(可能)致病变异位于剪接供体/受体±1,2二核苷酸之外或之内以及两者的相对位置(图6)。
注意:PS1的使用前提是,待分类变异的生信预测分数≥已知致病变异的生信预测分数。

图6
PART 8
获取与变异位置、剪接预测、剪接检测数据和变异类型有关的证据
剪接小组提供了基于变异位置、生信预测、剪接检测和已知致病变异确定适用证据的决策树(图7)。

图7
总结
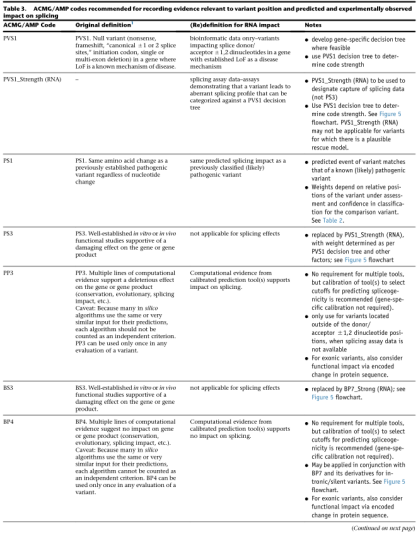
1.PVS1
已知LoF致病机制的基因中的无效变异(如无义、移码、±1或2剪接位点、起始密码子、单个或多外显子缺失);
更新后建议:仅有生信预测结果,已确定LOF为致病机制的经典剪接位点(±1,2)变异;对于剪接检测实验证明异常剪接的,应使用PVS1_Strength (RNA)。
2.PS1
与先前已确定的致病变异有相同的氨基酸改变;
更新后建议:变异预测的剪接效应与先前(可能)致病变异相同。
3.PS3
体外或体内功能研究支持变异对基因或基因产物具有破坏性作用;
更新后建议:不适用于体外剪接实验。
4.PP3
多种计算机软件预测显示变异对基因或基因产物产生有害影响(保守、进化、剪接影响等);
更新后建议:不需要多种工具,但建议对预测工具进行校准;当没有剪接实验数据时,仅用于非典型剪接位点变异;对于外显子变异,还应考虑变异对蛋白功能的影响。
5.BS3
体外或体内功能研究不支持变异对基因或基因产物具有破坏性作用;
更新后建议:不适用于体外剪接实验数据。
6.BP4
多种计算机软件预测变异不会对基因或基因产物产生有害影响(保守、进化、剪接影响等);
更新后建议:不需要多个工具,但建议对预测工具进行校准;选择恰当的剪接预测的阈值;对于外显子变异,还应考虑变异对蛋白功能的影响。
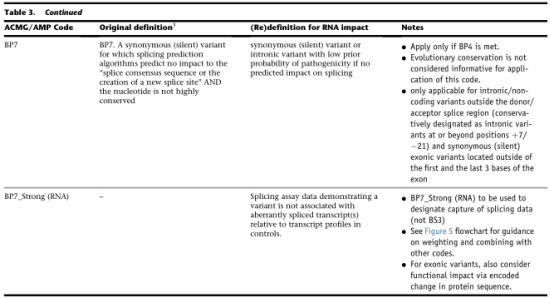
7.BP7
同义变异,其剪接预测不会对剪接标准序列产生影响,也不会产生新的剪接位点,而且它的核苷酸不高度保守;
更新后建议:只有满足BP4时才适用;不考虑变异位点的进化保守性;仅适用于指定剪接区域外的内含子/非编码变异(+7/-21之外的内含子变异或不位于外显子第1和最后3个碱基之外的同义外显子变异);剪接实验数据表明,变异不会引起异常剪接效应。适用BP7_Strong (RNA),不用BS3;对于外显子变异,还应考虑变异对蛋白功能的影响。
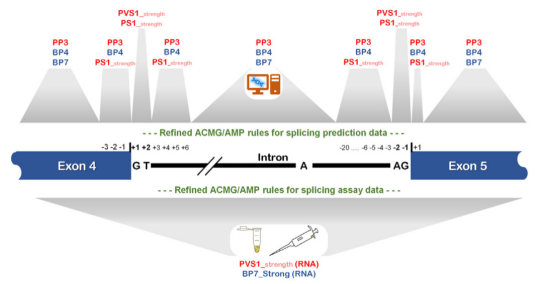
汇总图
