学术资源
应用ONT靶向测序平台对短串联重复序列扩增引起的疾病进行全面的基因诊断
短串联重复序列(short tandem repeat, STR)通常由2 ~ 6个碱基对组成,在基因组的特定位置以连续、重复的状态存在; STR约占人类基因组序列的7%,具有高度多态性。STR 的异常扩增是人类群体中一类重要的致病性变异。到目前为止,超过40个基因的STR扩增已经被证明会导致遗传性疾病,其中大多数表现为原发性神经或神经肌肉症状相关的疾病。
![1715323726022008019.jpg 6813 [转换]-01.jpg](/ueditor/image/20240510/1715323726022008019.jpg)
原发性神经或神经肌肉症状相关的疾病。包括亨廷顿舞蹈病(Huntington’s disease,HD;HTT)、脆性X综合征(fragile X syndrome,FXS;FMR1)、遗传性小脑共济失调(hereditary cerebellar ataxias ;RFC1、FXN等)、强直性肌营养不良(myotonic dystrophies ,DM;DMPK、CNBP)、肌阵挛性癫痫(CSTB, SAMD12, STARD7等)以及 额颞叶痴呆和/或肌萎缩侧索硬化症1型(C9orf72 frontotemporal dementia and/or amyotrophic lateral sclerosis ,C9orf72-FTD/ALS;C9orf72)等等。

由于STR扩增引起的相关疾病的患病率总体上较高,涉及的疾病基因数量多,重复扩增的大小也存在多样化,新基因不断被鉴定等因素,临床上对STRs的诊断需求日益增长。目前已有的分子诊断技术包括southern blot以及repeat-primed polymerase chain reaction (RP-PCR)劳动密集且不精确,需要单独用特异性引物或探针检测不同的STR。
二代测序作为短读长测序技术亦不适合重复序列的检测。长读长测序包括Oxford Nanopore Technologies (ONT) and Pacific Biosciences可以准确检测大片段和复杂STR扩增,但是全基因组测序费用昂贵。ONT的“ReadUntil”功能可以通过参数设置使纳米孔接受或拒绝特定的DNA序列片段,从而达到靶向测序的目的,这一功能很有可能实现对已知的所有致病STR进行简单、有效的检测,但还没有经过验证。
研究内容
本文以与神经系统疾病相关的致病性STR位点的37个基因、基因型-药物相关的PGx基因28个以及ACMG指南推荐的临床相关的59个致病基因为靶向测序目标,同时包含上述基因±50kb的侧翼基因序列,以确保启动子和UTR区域等其它局部调控元件的覆盖,采用Readfish软件的ONT ReadUntil API功能对非靶向测序目标基因进行排除从而达到靶向测序的目的。
研究方法及技术路线
本研究样本选用Coriell标准品,以PR-PCR或者southern blot(大片段STR扩增)技术检测结果为对照,应用ONT ReadUntil 技术进行靶向测序,包括37个STR基因、28个药物代谢基因以及59个其它遗传病相关基因(图1)。
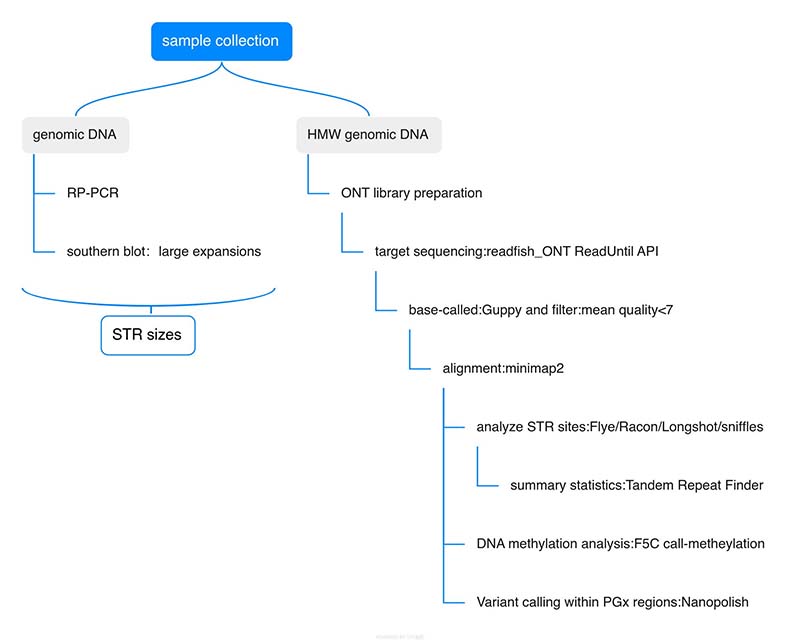
图1.ONT靶向测序技术路线图
研究结果
实验目标测序基因序列约50.5Mb,占人类参考基因组序列(hg38)的1.6%,包含37个与神经系统疾病相关的致病性STR基因、28个药物代谢基因和59个ACMG指南推荐其它遗传疾病相关基因以及上述基因±50kb侧翼基因序列。通过Readfish软件参数设置可以达到靶向测序的目的,最终目标区域的平均读取深度比非目标区域高4.6×,平均覆盖率约高9-40×(图2)。
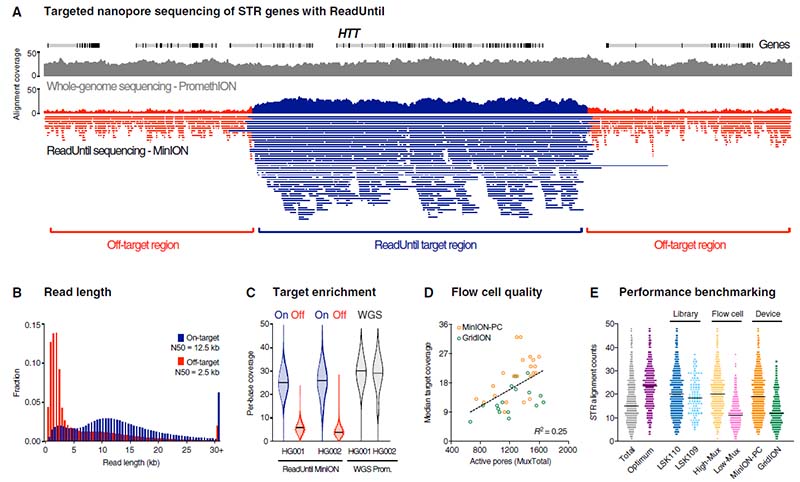
图2.ONT ReadUntil针对致病性STR位点的靶向测序结果图
A.HTT基因全基因组测序覆盖度与靶向测序覆盖度对比图;B.靶向测序的目标区域与非目标区域reads长度对比图;C.靶向测序目标基因序列覆盖度与非目标区域覆盖度以及WGS覆盖度的对比图;D.MinION-PC和GridION运行ReadUntil功能流动池上起始活性孔数量与平均覆盖度的关系的对比;E.建库试剂、流动池、测序设备的对比图
①STR靶向ONT测序结果
选择神经系统相关疾病的患者25例验证靶向纳米孔测序的有效性和实用性,包括5例HD、2例FXS、6例CANVAS、1例SBMA、5例DM1、1例NIID、2例FRDA、1例ALS、1例SCA1 、1例OPMD以及6例已知的前突变携带者和6例未患病个体,共37例样本;上述样本已通过RP-PCR或southern blot等临床分子诊断技术验证。靶向ONT测序结果显示:HD受累患者5例,未发现HTT基因致病性扩展32例,与RP-PCR或Southern blot检测结果相关性分析显示R2=0.996(图3);FXS男性患者2例,1例女性携带者来自正常个体和1例男性前突变STR扩展,与RP-PCR或Southern blot检测结果相关性分析显示R2=0.993(图3)。
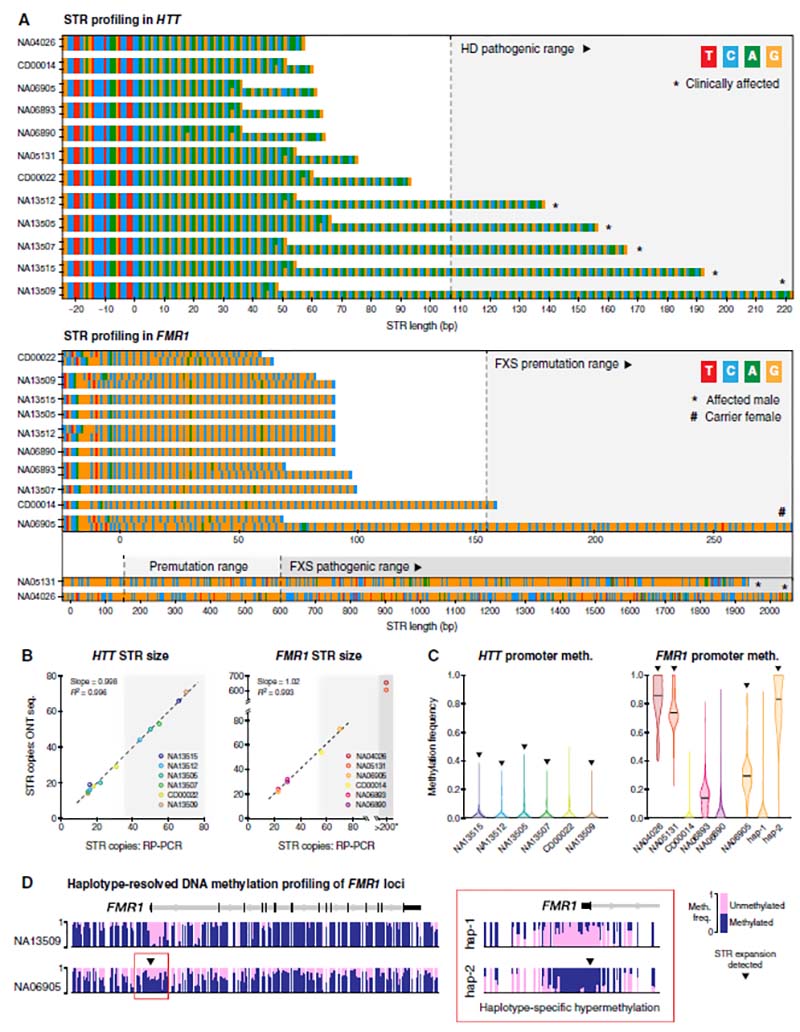
图3.HTT和FMR1 STR位点单倍体组装和DNA甲基化分析
A.Coriell参考DNA样本的HTT和FMR1基因的STR测序结果图,男性个体FMR1只有一个拷贝;B.ONT靶向测序和RP-PCR检测HTT和FMR1 STR大小相关性分析,有2例样本的FMR1 STR大小超过了RP-PCR的检测上线;C.HTT和FMR1 启动子区域内DNA甲基化频率分布,“▼”代表有致病扩展;D. 基因组浏览器视图显示了两个样本NA13509(无STR扩增的女性) 和NA06905 (FMR1前突变女性携带者)的完整FMR1基因座的DNA甲基化特征。插页显示NA06905的单倍型特异性启动子甲基化。
CANVAS患者中检出6例,其它未受影响的个体中没有检出相关5碱基重复扩增,与RP-PCR或Southern blot检测结果相关性分析显示R2=0.946(图4);其余STR相关疾病患者均有检出,与预期一致。
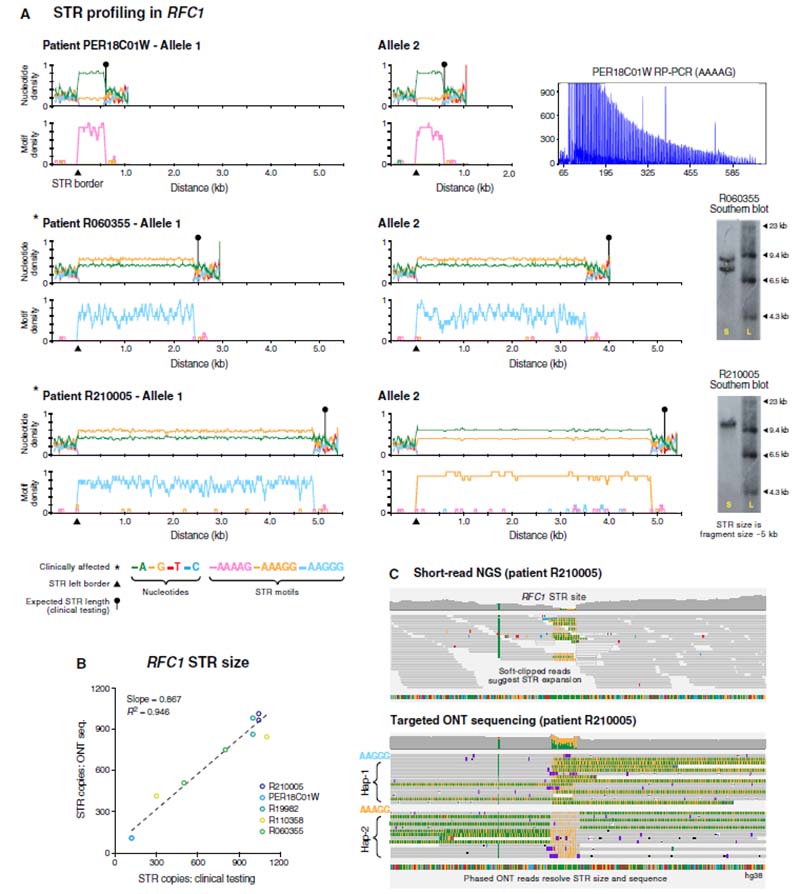
图4.RFC1基因致病性STR位点的单倍型组装分析
A.3例样本RFC1动态突变长度与基因频率图,右边对应的是其RP-PCR或Southern blot检测结果图;B.ONT测序结果与其它临床检测方法检测结果相关性分析显示R2=0.946;C.NGS测序结果只提示STR扩增但不能确定具体大小,而靶向ONT测序结果直接给出STR扩增大小。
②PGx基因检测结果
许多药物代谢基因如CYP2D6等与许多神经系统疾病的药物相关;而这些基因大多具有高度多态性,存在高度同源性假基因,并且经常包含结构变异等,在采用短读长测序技术检测其变异存在极大的困难。因此文献中将28个PGx基因纳入目标基因,无需增加额外成本,可以灵活选择或者排除相关基因。两个参考样本HG001和HG002的测序结果显示,SNVs的灵敏度分别是95%、97%,精确度分别是96%、98%;但是indel变异类型的灵敏度仅36%和56%,精确度仅56%和58%。因此indel变异类型在ONT测序平台检测的可靠性不高(图5)。
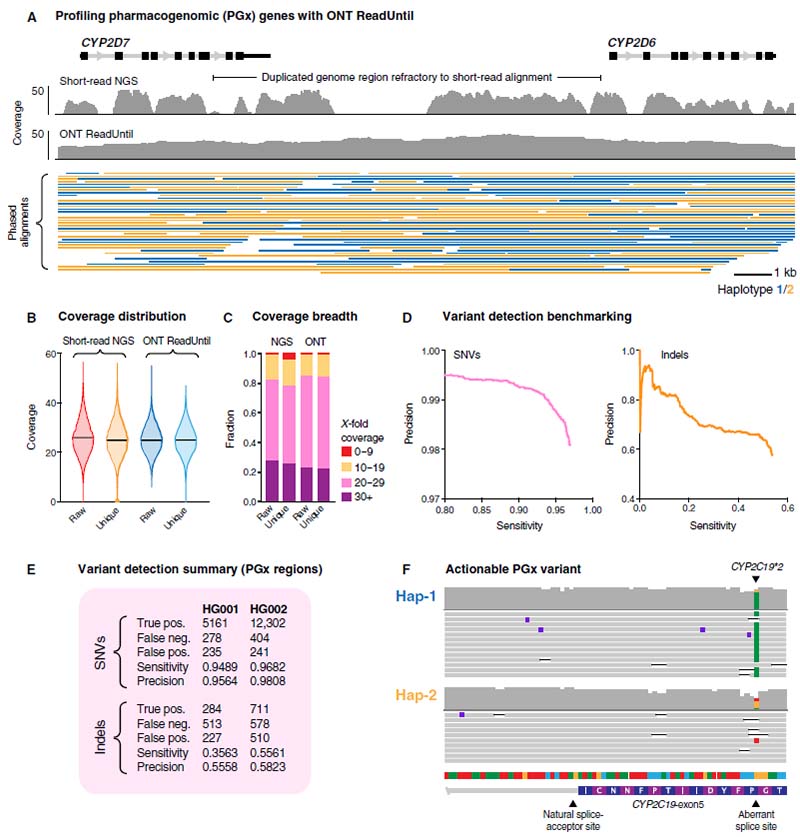
图5.PGx基因靶向ONT测序结果及分析图
A.HG001样本的CYP2D6及其邻近基因CYP2D7在短读长NGS_illumina测序平台和长读长ONT ReadUntil测序平台覆盖率分布对比图;B小提琴图显示.短读长NGS与靶向ONT技术目标基因覆盖度,包括原始和校准后的对比图;C. 条形图显示了不同测序深度覆盖的PGx目标区域的比例:红色,0 ~ 9×;黄色,10 ~ 19×;粉色,20 ~ 29×;紫色,≥30×;D.SNV、Indels变异类型的灵敏度与精确度;E.显示PGx目标内最终变异检测统计数据的汇总表,插入缺失准确度较差,但SNVs检测具有较高的灵敏度和精密度;F.显示了在HG001中使用ONT ReadUntil检测到的临床可操作的PGx等位基因(CYP2C19*2)的一个例子。
结论
本研究实验证明应用ONT ReadUntil靶向测序技术检测神经系统相关疾病的STR扩增完全是可行的,克服了短读长测序技术检测STR扩增存在剪辑偏好性、重复数不准确以及大片段重复序列无法确定大小的技术难点;同时还可以对致病性STR扩增序列进行甲基化分析,进一步了解致病性DNA局部的甲基化特征;进一步可以增加药物代谢基因组测序,可以为神经系统相关疾病的用药进行科学的指导。
