学术资源
Science:人类和灵长类动物中可耐受的遗传变异图谱

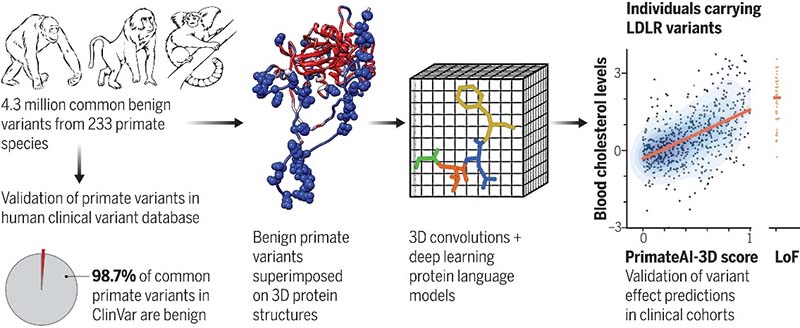
2023年6月2日,Science在线发表了Illumina人工智能实验室和多个课题组合作的研究成果“The landscape of tolerated genetic variation in humans and primates”。研究建立了包含430万个常见错义变异的数据集,并将其与深度学习模型PrimateAI-3D结合,在识别预测意义不明的变异并识别其致病性方面具有重要意义。
一、研究背景
①所要解决的问题:由于大量临床意义不明的基因变异的存在(~99.9%)以及大多数人类遗传变异的罕见性,通过基因检测通常难以得到明确诊断。
②改进的策略:利用遗传信息高度相似的灵长类动物的数据(黑猩猩与人类的蛋白序列同一性达99.4%),系统地对同源变异分类并研究这些变异在人类中的致病性。
③局限性:可用的灵长类种群测序数据集非常小,限制了发现的常见变异的数量以及可训练的机器学习模型的规模。
二、研究结果
①建立包含灵长类动物谱系中430万个良性错义变异的数据库
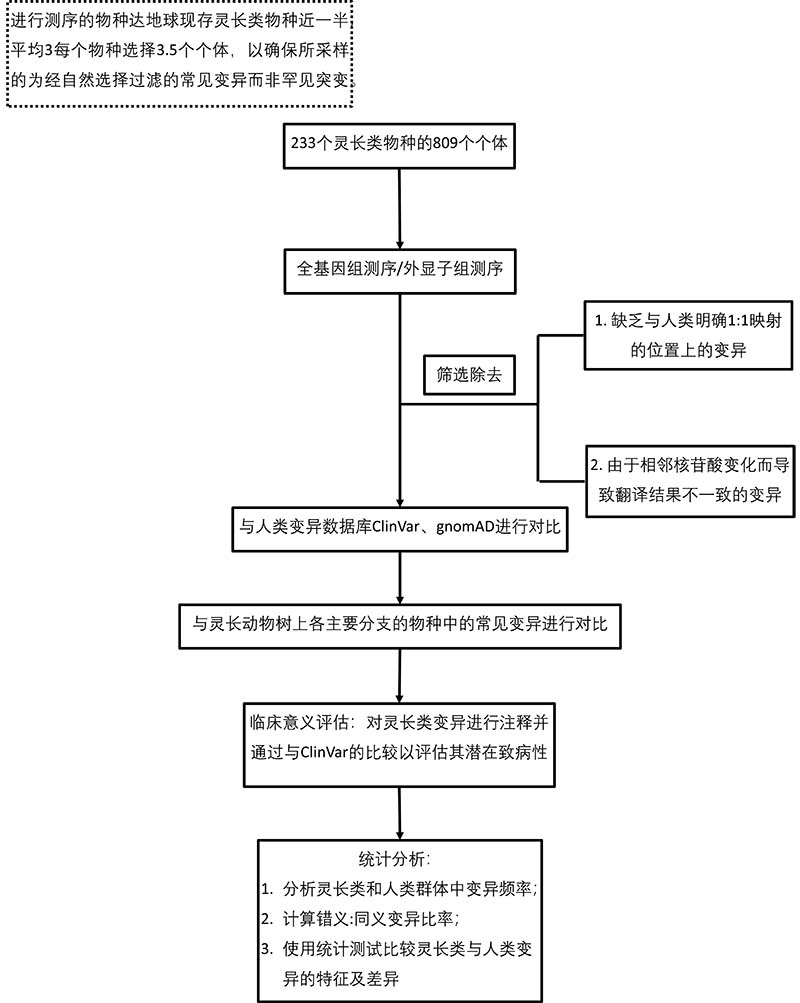
主要发现:
1)灵长类动物中的外显子组变异频率高。
灵长类和人类变异之间的重叠度较低,表明每个物种都有独立的突变起源;并且值得注意的是,尽管测序的个体数量少的多,但灵长类动物群体中包含的外显子组变异比gnomAD群多约20%,突出了非人类灵长类物种的遗传多样性。
2)灵长类动物中发现的错义突变绝大部分为良性/可能良性。
与同义突变相比,灵长类动物群体中的错义突变明显较少;且灵长类动物错义变异在ClinVar数据库中因其良性后果而大量富集,表明它们致病性的概率较低(~99%良性/可能良性)。
3)致病变异频率最高的人类疾病基因区域往往在灵长类动物中具有较少的常见变异,表明人类和灵长类动物经历相似的选择压力。
通过与gnomAD的对比分析,发现gnomAD队列中包含的致病变异比例更高(自然选择在消除灵长类动物的有害变异方面更有效),这可能是因为人类人口数的急剧增长使得随机的新发变异大大增多而自然选择尚未有足够时间来清除有害的变异。少数人类致病性变异在灵长类动物
4)随着物种之间进化距离的增加,利用一个物种中的常见变异来预测另一物种中变异的致病性的可靠性降低。
研究人员研究了灵长类动物树的不同分支以及哺乳动物、鸟类和鱼类的变异。研究发现,灵长类动物(甚至是远亲物种)中的常见变异对人类大多是良性的;而在进化距离更远的物种中(如有胎盘哺乳动物和鸡),变异对人类良性的比例下降。
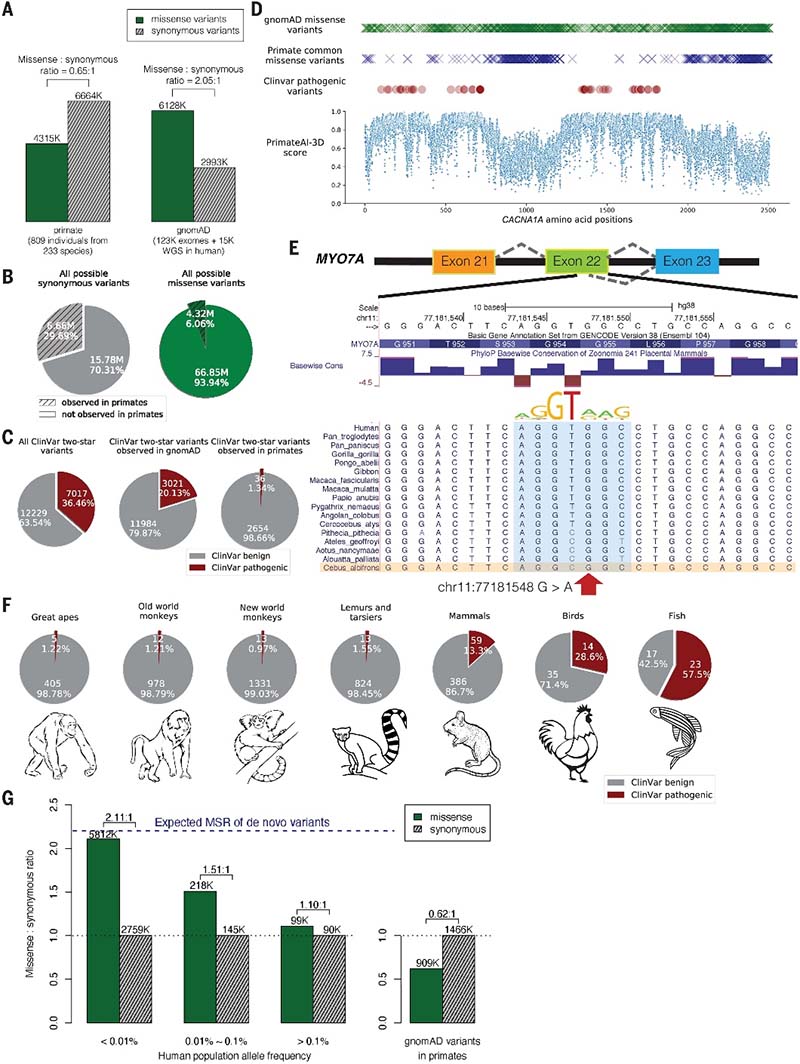
图1.灵长类动物中的常见变异对人类大多是良性的
②人类与非人灵长类动物中的基因选择约束
接下来研究人员着手确定人类与其他灵长类动物相比经历了不同自然选择压力的基因,以了解人类特有的进化中的适应。他们主要采取的研究策略如下图:
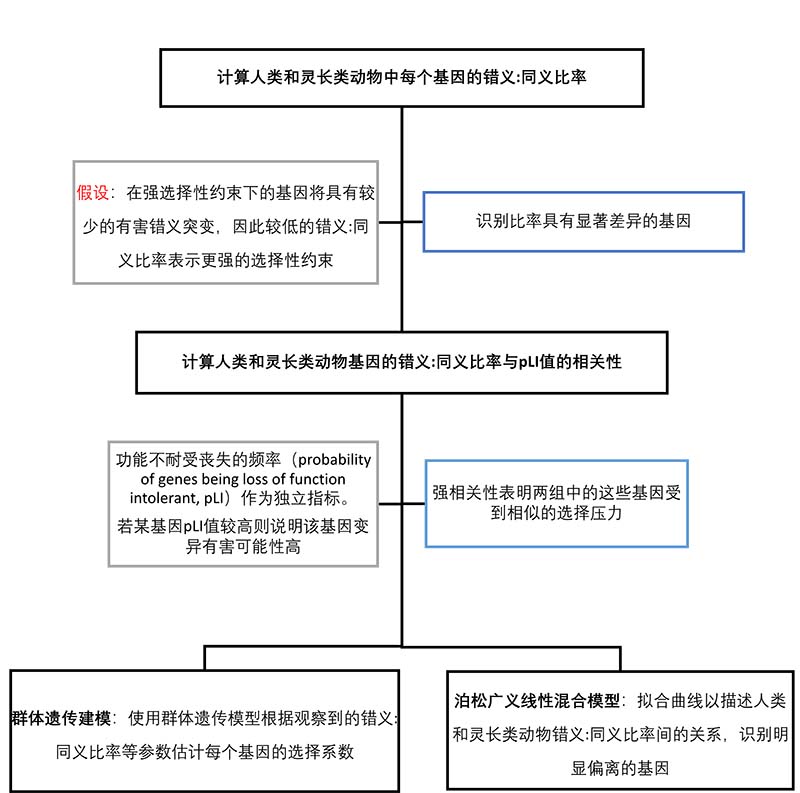
使用两种方法识别出的结果基本相同。最终,他们共发现了39个在人类与其他灵长类动物之间存在不同选择压力的基因。其中,CFTR、GJB2和CD36在人类中受到的选择减少,它们分别是囊性纤维化(cystic fibrosis)、遗传性耳聋(hereditary deafness)及血小板糖蛋白缺乏症(platelet glycoprotein deficiency)的常染色体隐性致病基因;这可能是由于杂合子优势可以抵御特定环境中的病原体。而TERT相较于其他灵长类基因在人类中受到更多的选择,这是一个与维持端粒长度有关的基因,可能适应人类相较于其他灵长类寿命的延长。但是,受到当前较小数据集的限制,需要有更大、更多样化的群体来支持人类与其他灵长类动物谱系中关于受到不同选择压力的基因的结论。
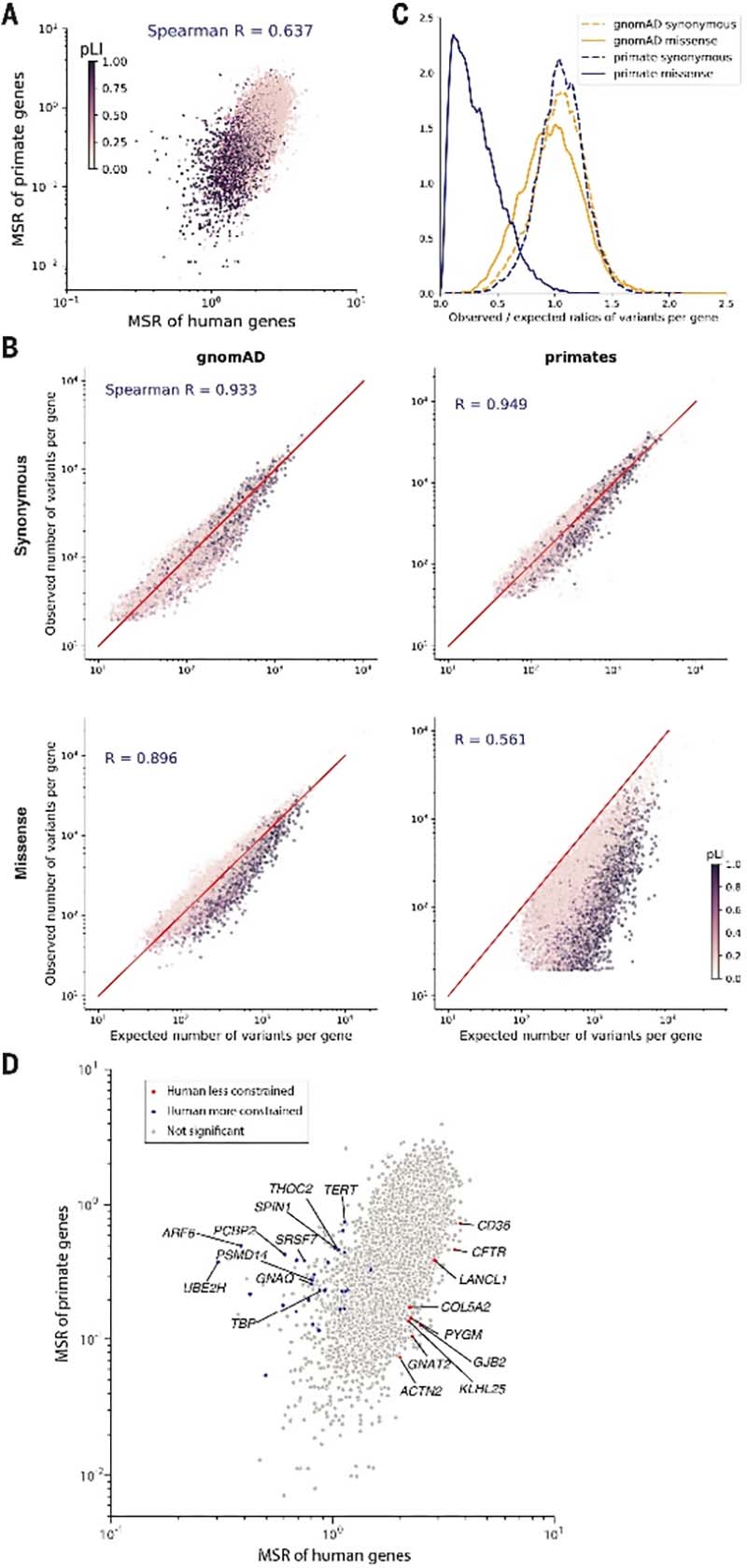
图2.与人类相比其他灵长类动物中的基因选择约束。A.灵长类动物和人类基因之间的错义同义突变比率的散点图。每个基因都由其pLI分数着色,颜色较深的点显示单倍计量不足基因;C.所有基因的同义(虚线)和错义(实线)变体的观察/预期比率的分布(灵长类动物:橙;gnomAD基因:蓝);D.错义散点图:灵长类动物和人类基因之间的同义比率,灰点显示不显著的基因。
③PrimateAI-3D,一种用于对蛋白质变化变异进行分类的深度学习网络
PrimateAI-3D是一种用于变异致病性预测的半监督3D卷积神经网络(semisupervised 3D convolutional neural network)模型。研究人员使用450万个可能产生良性后果的常见错义变异对其进行了训练,数据涵盖了gnomAD、TOPMed和UK Biobank中的233个非人灵长类物种及人类的常见错义变异,占所有可能的人类错义变异的6.34%,极大扩大了可用于机器学习方法的训练数据集。与以往在线性序列上的深度学习模型不同,他们以2Å的分辨率对蛋白质的3D结构进行体素化(voxelized)并利用3D卷积使该网络能够识别仅从序列中可能不明显的关键结构区域。研究人员使用了实验来源及计算预测(AlphaFold DB及HHpred)的蛋白质结构以最广泛地覆盖人类基因。
值得注意的是,该网络并未接受人为设计的或临床变异数据库中注释变异的训练,从而避免了变异注释中潜在的人类偏见;而是仅将蛋白质的多序列比对和3D结构作为输入,学习根据常见灵长类动物变异的局部富集/缺失来推断致病性。
由于上述训练数据集仅包含良性变异,研究人员创建了一组随机选择的变异对照,训练PrimateAI-3D将常见变异从匹配的对照组中分离;同时,他们还训练了两个模型(三维卷积“填空”模型与transformer架构)来预测缺失的氨基酸,并将其作为额外的损失函数以进一步优化PrimateAI-3D的预测结果。接下来研究人员对Primate-AI-3D进行了性能评估并与其他15个机器学习模型在6个维度进行了比较:
1)在大规模的临床患者队列(UKBB中200643名个体)中预测罕见错义变异对定量临床表型的影响;
2)区分神经发育障碍(DDD)患者与健康对照中出现的错义新发变异(DNM);
3)区分自闭症谱系障碍(ASD)患者与健康对照中的DNM;
4)区分先天性心脏病(CHD)患者与健康对照中出现的DNM;
5)区分ClinVar中注释的良性及致病变异;
6)评估PimateAI-3D与9个基因的体外深度突变扫描(deep mutational scan,DMS)实验检测的平均相关性。
结果显示,PrimateAI-3D在所有临床基准测试中的表现均优于其他分类器,特别是在区分致病和良性变异方面;它能够在临床应用中准确预测蛋白质变异的致病性,具有广泛的应用前景。
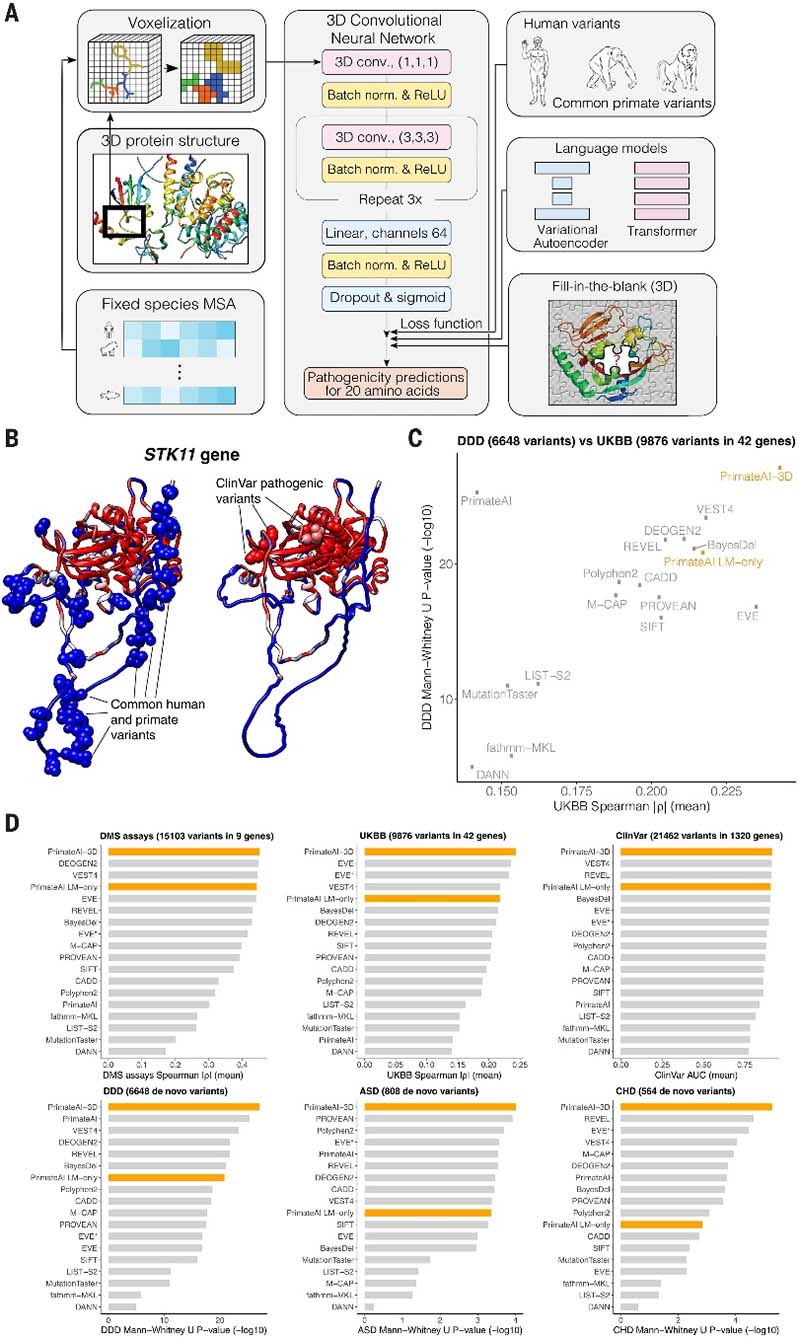
图3.PrimateAI-3D的体系结构和变异分类性能。A.PrimateAI-3D工作流程;B.STK11基因的蛋白质结构,由PrimateAI-3D致病性预测评分着色(蓝色:良性;红色:致病性);C.显示在两个临床基准(DDD和UKBB)中预测错义变异致病性的方法的性能的散点图;D.检测了6 个数据集(DMS分析、UKBB、ClinVar、DDD、ASD 和 CHD)的不同方法的性能。
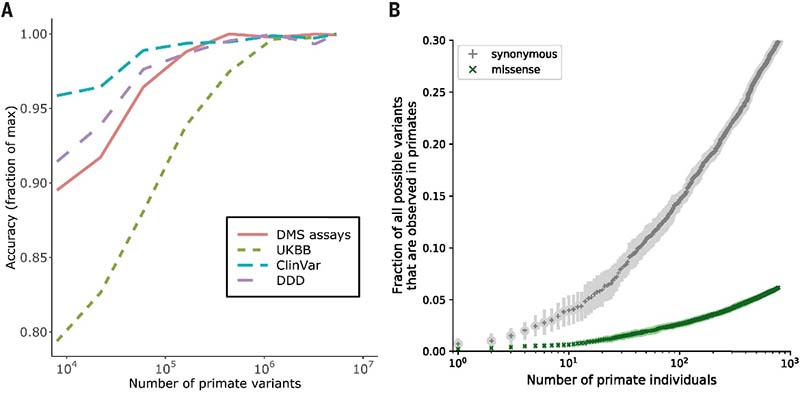
图4.训练数据集大小对分类精度的影响。A.随着训练数据集中常见灵长类变异数量(x轴)的增加,PrimateAI-3D的性能得到改善
④神经发育障碍的候选疾病基因的发现
新发变异在人群中的发生频率高,但致病频率低、经受的选择压力较小,因此成为散发性、复杂性疾病的成因之一。新发变异在神经发育障碍中发挥重要作用,准确识别它们的潜在致病性有助于此类疾病的早期诊断与治疗。
研究人员利用PrimateAI-3D来提高发现候选疾病基因的能力,这些基因在神经发育障碍队列中富含致病性新发变异。根据三核苷酸上下文对背景突变率的估计, DDD队列中受影响个体的新发错义突变比预期高了1.36倍。研究人员将PrimateAI-3D的分类阈值设为0.821,可找出与该队列中新生错义变异数量相同的致病性错义变异(n=7,238)。按此阈值对错义变异进行分层可将致病性新发错义变异的富集度增至2倍,从而大大提高了队列中疾病基因的发现能力。通过应用PrimateAI-3D对致病性错义变体进行排序,在全基因组范围内鉴定了290个与智力障碍相关的基因,其中272个是之前已经发现的基因。BMPR2和RYR1基因已被明确注释为与神经表型无关,剩余候选基因需经独立验证并了解它们的表型效应范围。
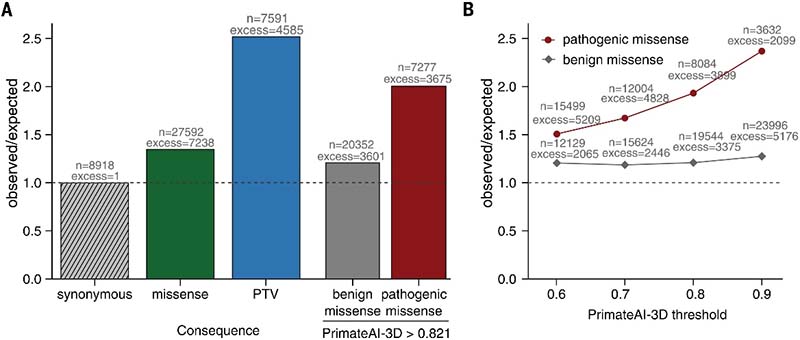
图5.在不同的PrimateAI-3D阈值下,良性和致病性错义的富集高于预期
三、研究总结
本研究主要呈现了一种灵长类动物群体测序数据与深度学习模型的成功结合,有助于理解意义不明确的变异。灵长类动物群体测序和大规模人类测序发挥着互补作用,灵长类动物测序可以为训练深度学习模型提供丰富的可能良性变异来源,而人类测序则识别出具有潜在表型效应的稀有变异。利用灵长类动物常见变异训练的分类器有助于区分良性和致病性罕见变异,有助于发现新的基因-表型关系。灵长类动物变异作为一种资源,与深度学习模型的结合帮助提高了在多个临床基准测试中对变异效应的预测准确性,对人类遗传学的进一步探索至关重要。因此,我们必须保护珍贵的遗传多样性,利用它们的基因组以更好地理解人类自身。
